티스토리 뷰
728x90
단백질의 구조
- 단백질:
- 아미노산의 염기 서열에서 형성된 생체고분자(polypeptide), 아미노산 -> 축합반응 -> 단백질 (보통 아미노산 30개 이상이 단백질)
- 단백질은 다수의 비공유적 상호작용(수소/이온/반데르발스/소수성 결합 등)에 의해 구동되는 1 이상의 특정한 공간적 형태(folding)
- 단백질 기능의 이해는 이의 3차원 구조를 결정하는 것이 중요함
- 단백질 구조:
- 아미노산 사슬에 있는 원자와 분자의 3차원 배열
- 수만 ~ 수천 개의 아미노산 크기(1~100 nm)
- 단백질의 4가지 구조
- 1차 구조

- 폴리펩타이드 사슬에서 아미노산 서열 (펩타이드 결합)
- 단백질에 해당하는 유전자에 의해 결정
- 단백질 서열은 그 단백질에 고유하며, 단백질의 구조 및 기능 정의
- 질량 분석법이나 유전 부호를 사용하여 단백질 순서를 알 수 있음
- 펩타이드 결합시 불분자가 소실되어 단백질이 아미노기로 구성
- 인산화 등, 번역 후 변형이 보통 1차 구조로 간주되며, 유전자에서 읽을 수 없음
- 2차 구조
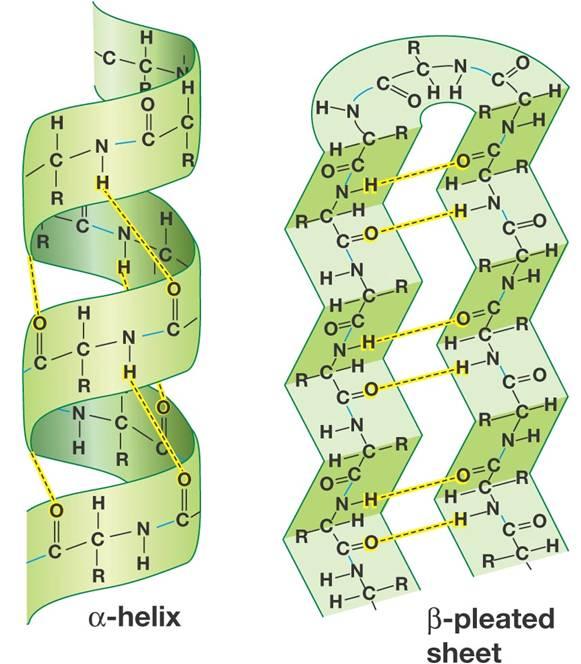
- 폴리펩타이드 골격 사슬 상의 매우 규칙적인 국소 구조 (by 수소 결합 패턴)
- 알파 나선 구조, 베타 병풍 구조 모두 펩타이드 골격에서 모든 수소 결합 공여체 및 수용체를 포화시키는 방법
- 3차 구조
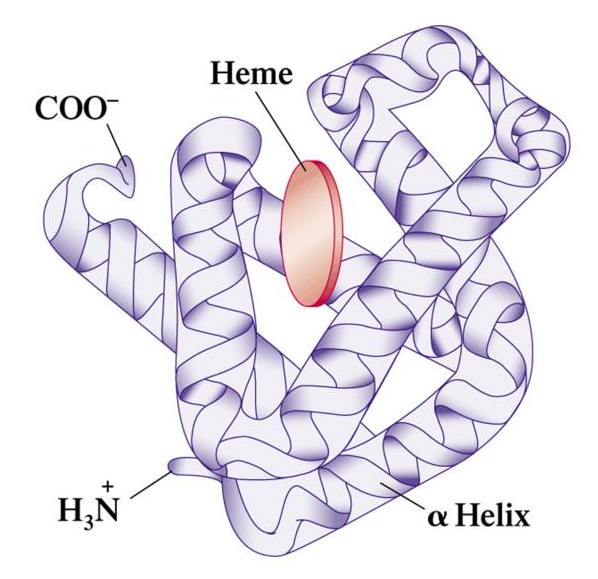
- 단백질의 3차원 구조
- 2차원 구조가 구형(globular) 구조로 접혀 있음
- 4차 구조
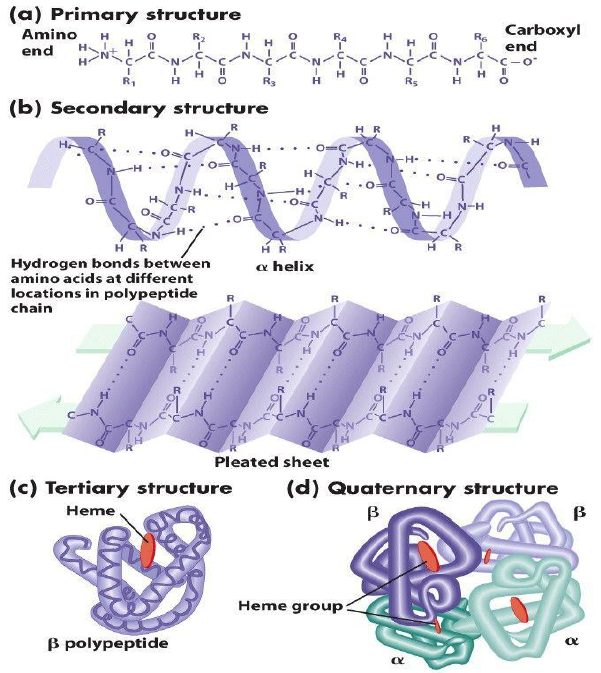
- 단일 기능 단위(다량체)로서 작용하는 둘 이상의 개별 폴리펩타이드 사슬(서브 유닛)로 구성된 3차원 구조
- 단백질 접힘 (protein folding)
- 나선 다발 구조(Helix bundle), 베타 통 구조(Beta barrel), 로즈만 접힘 또는 단백질의 구조적 분류 데이터베이스에 제공된 다른 접힘과 같은 일반적인 단백질 구조
- 단백질 구조 결정
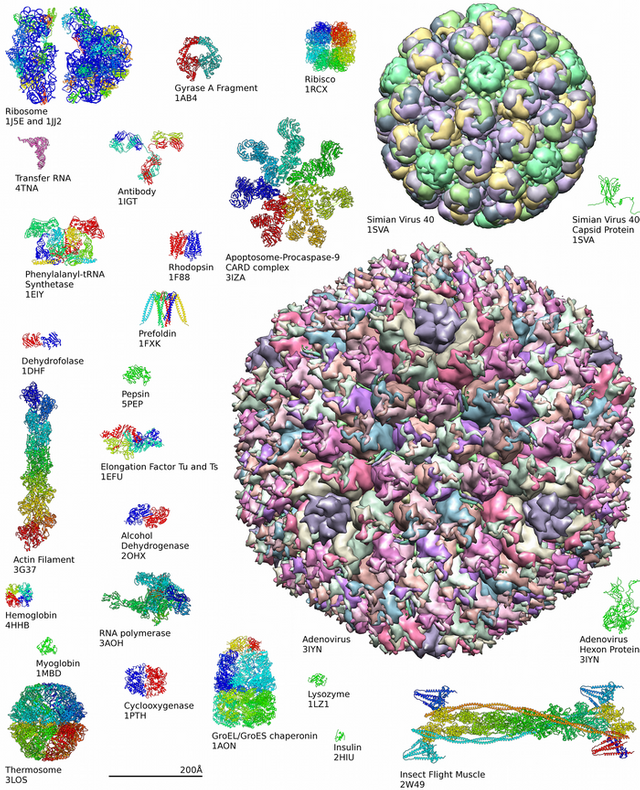
- Protein Data Bank(PDB)에서 사용할 수 있는 단백질 구조
- 결정화된 상태에서 단백질 내 전자의 3차원 밀도 측정 가능 -> 모든 원자의 3차원 좌표를 유추할 수 있음
'데이터 분석 > 생물 및 의료 데이터' 카테고리의 다른 글
| [유전체] Imputation (0) | 2023.01.04 |
|---|---|
| [유전체] prob ID를 RS ID로 바꾸기 (0) | 2023.01.04 |
| 우분투에 PLINK 설치하기 (0) | 2023.01.04 |
| 코로나 바이러스 유전자와 단백질 구조 (0) | 2023.01.04 |
| NGS data 활용 영역 (0) | 2023.01.04 |
댓글
공지사항
최근에 올라온 글
최근에 달린 댓글
- Total
- Today
- Yesterday
링크
TAG
- ECG
- pre-train
- 생존분석
- r
- cnn
- sequenced data
- vcf
- 생존함수
- gray2rgb
- rgb2gray
- GradCam
- GPU설치
- Bioinfo
- plink
- NGS
- 딥러닝
- fasta
- 인공지능
- psychopy
- sounddevice
- SNP
- 평균분석
- pmm
- 생존곡선
- PTB
- featuremap
- 그룹비교
- HRV
- 실험통계
- missing_value
| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
글 보관함
